网站维护
系统内容更新/升级中

2020/12/21
近日,药明康德HitS团队在Organic Letters杂志上以封面文章发表了可见光介导的脱羧偶联反应,在DNA编码化合物库(DNA-Encoded Library,以下简称DEL)中实现了1,2-氨基醇化合物的高效合成[1]。该方法采用α-氨基酸和羰基化合物分别作为亲核部分和亲电部分,实现了光氧化还原介导的sp3碳碳键偶联反应,具有条件温和、高效且底物适用性广等特点,未来可实现百万级化合物的合成,在DEL库的构建中具有巨大的应用前景。
DEL技术与DNA兼容的化学反应
DEL技术,作为新一代的高通量筛选技术,将化学合成与基因编码策略有机地结合起来,在新药研发中得到了广泛应用。在合成DNA编码化合物库的过程中,最大挑战之一就是DEL分子中的DNA部分限制了可用的有机化学反应类型,很大程度影响了化合物库的化学空间多样性。因此,开发DNA兼容的化学反应,是构建具有多样性化合物库的关键。目前,DNA兼容的化学反应,在C-N键(酰胺/脲化合物合成、还原胺化、Michael加成、Buchwald偶联反应、SNAr、SN2等)构建方面取得了一系列的成功;但在C-C键构建方面局限于sp2碳碳键(Suzuki偶联反应、Wittig反应等),而广泛存在的C(sp3)-C(sp2)与C(sp3)-C(sp3)构建则很少报道。
值得关注的是,过去的十几年里,与可见光介导的有机化学反应相关的报道呈指数型增长。与传统的合成方法相比,可见光介导的有机反应具有反应条件温和、步骤简单和环境友好等优点。同时,在C(sp3)-C(sp2)与C(sp3)-C(sp3)构建上,可见光催化体系展现出了优异的催化合成价值和应用潜力,广泛应用于复杂化合物的合成中[2]。因此,如果能将可见光介导的光化学反应成功应用于DEL合成中,将极大地提升化合物库的化学空间多样性。

DNA兼容的可见光介导的化学反应的发展
近年来,化学家们也开发出了DNA兼容的可见光介导的化学反应,并将它们应用于DNA编码化合物库的构建(图1)。
2019年,宾夕法尼亚大学Gary A. Molander课题组将可见光介导的有机光化学反应应用到DNA编码化合物库的合成,将该领域的研究推向了一个新的高度。他们使用镍/光氧化还原双催化体系,实现了芳基卤代物与烷基自由基前体的C(sp3)-C(sp2)交叉偶联反应[3]。同时,辉瑞制药(Pfizer)也开发了DNA兼容的可见光介导的Giese反应,用于C(sp3)-C(sp3)构建[4]。鉴于1,2-氨基醇是各种天然产物和药物活性分子的重要单元,药明康德HitS团队开发出了DNA兼容的可见光介导的α-氨基酸与羰基化合物的脱羧偶联反应,用于合成1,2-氨基醇化合物[1]。

可见光介导的脱羧偶联反应条件温和且高效
该反应使用[Ir(ppy)2(dtbbpy)]PF6作为光催化剂,在室温条件下,反应10分钟即可高效得到目标产物。首先,使用a-氨基酸2a作为自由基受体,本文考察了带有不同官能团的DNA连接的羰基化合物1对反应的影响(图2)。该方法很好地适用于芳香醛类衍生物,绝大多数取代基(例如,烷基、芳基、卤素、三氟甲基、酰胺、含N/O/S杂环等)的位置对反应的影响较小,都能以良好到优秀的收率得到目标产物。此外,该方法也较好地适用于芳基-烷基酮、二芳基酮与二烷基酮等底物,只是相对于芳基醛化合物,收率会有所降低。
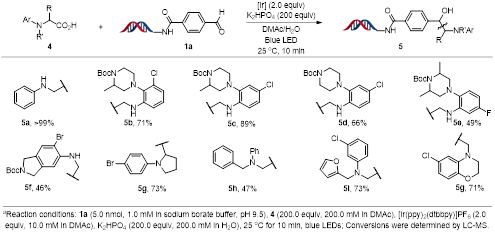
随后,作者也对a-氨基酸4进行了扩展,如图3所示,多种多官能团(胺基、Cl、Br等)的a-氨基酸,都能和相应的on-DNA苯甲醛发生脱羧偶联反应。


为了满足DNA编码化合物库的生产需求,团队也自主设计了可用于高通量生产的光反应装置(图4)。该反应装置可以对反应温度、光照强度等精准调控,一次性可完成12个96孔板的生产工作。
此外,团队也进行了相关的实验,用来评估光反应对DNA的损伤情况。DNA连接实验与二代测序实验结果表明,可见光介导的脱羧偶联反应,具有DNA兼容性,不会对DNA造成明显损伤。因此,团队也将该反应应用到了高质量DNA编码化合物库的构建中。
总结
综上,该工作拓展了适用于DEL的可见光介导的脱羧偶联反应。在温和的条件下,使用a-氨基酸和羰基化合物作为底物,实现了1,2-氨基醇的高效合成,并成功地将其运用到DNA编码化合物库的构建中。
参考文献:
(1) Wen, Huanan; Ge, Rui; Qu, Yi; Sun, Jialin; Shi, Xiaodong; Cui, Weiren; Yan, Hao; Zhang, Qi; An, Yulong; Su, Wenji; Yang, Hongfang; Kuai, Letian; Satz, Alexander. L.; Peng, Xuanjia. Org. Lett. 2020, DOI:10.1021/acs.orglett.0c03461
(2) Romero, N. A.; Nicewicz, D. A. Chem. Rev. 2016, 116, 10075-10166.
(3) Phelan, J. P.; Lang, S. B.; Sim, J.; Berritt, S.; Peat, A. J.; Billings, K.; Fan, L.; Molander, G. A. J. Am. Chem. Soc. 2019, 141, 3723-3732.
(4) Kölmel, D. K.; Loach, R. P.; Knauber, T.; Flanagan, M. E. ChemMedChem 2018, 13, 2159-2165.